TO THE EDITOR:CRISPR (clustered regularly interspaced short palindromic repeats)-based diagnostic tests collectively provide a nascent platform for the detection of viral and bacterial pathogens. Methods such as SHERLOCK (specific high-sensitivity enzymatic reporter unlocking), which typically use a two-step process (target amplification followed by CRISPR-mediated nucleic acid detection), have been used to detect SARS-CoV-2. These approaches, however, are more complex than those used in point-of-care testing because they depend on an RNA extraction step and multiple liquid-handling steps that increase the risk of cross-contamination of samples.
Here, we describe a simple test for detection of SARS-CoV-2. The sensitivity of this test is similar to that of reverse-transcription-quantitative polymerase-chain-reaction (RT-qPCR) assays. STOP (SHERLOCK testing in one pot) is a streamlined assay that combines simplified extraction of viral RNA with isothermal amplification and CRISPR-mediated detection. This test can be performed at a single temperature in less than an hour and with minimal equipment.
The integration of isothermal amplification with CRISPR-mediated detection required the development of a common reaction buffer that could accommodate both steps. To amplify viral RNA, we chose reverse transcription followed by loop-mediated isothermal amplification (LAMP) because LAMP reagents are widely available and use defined buffers that are amenable to Cas enzymes. LAMP operates at 55 to 70°C and requires a thermostable Cas enzyme such as Cas12b from Alicyclobacillus acidiphilus (AapCas12b). We systematically evaluated multiple LAMP primer sets and AapCas12b guide RNAs (a guide RNA helps AapCas12b recognize and cut target DNA) to identify the best combination to target gene N, encoding the SARS-CoV-2 nucleocapsid protein, in a one-pot reaction mixture (see Figs. S1 through S3 in the Supplementary Appendix, available with the full text of this letter at NEJM.org). We termed this assay STOPCovid, version 1 (STOPCovid.v1). As expected, STOPCovid.v1 detection produced a signal only when the target was present, whereas LAMP alone can produce a nonspecific signal (Fig. S3E). STOPCovid.v1 is compatible with lateral-flow and fluorescence readouts and can detect an internal control with the use of a fluorescence readout (Figs. S4 through S6).
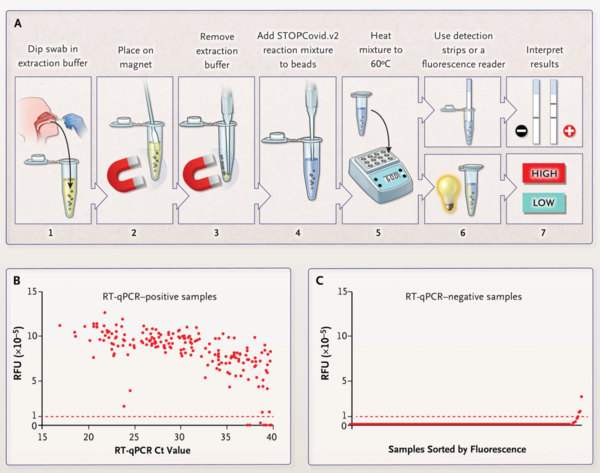
To simplify RNA extraction and to boost sensitivity, we adapted a magnetic bead purification method (Fig. S9). The magnetic beads concentrated SARS-CoV-2 RNA genomes from an entire nasopharyngeal or anterior nasal swab into one STOPCovid reaction mixture. We streamlined the test by combining the lysis and magnetic bead-binding steps and eliminating the ethanol wash and elution steps to reduce the duration of sample extraction to 15 minutes with minimal hands-on time. We refer to this streamlined test as STOPCovid, version 2 (STOPCovid.v2) (Figure 1A).
We compared STOPCovid.v2 with the Centers for Disease Control and Prevention (CDC) standard two-step test (i.e., RNA extraction followed by RT-qPCR) (Fig. S10C). The concentration of substrate by magnetic beads in STOPCovid.v2 allowed detection of viral RNA from the entire swab sample, yielding an input (in terms of quantity of viral RNA) that was 600 times that afforded by the CDC test. As a result, STOPCovid.v2 reliably detected a viral load that was one thirtieth that detected by the CDC RT-qPCR test (100 copies per sample, or 33 copies per milliliter, as compared with 1000 copies per milliliter). Analysis of two independent dilution series from nasopharyngeal swab samples revealed that STOPCovid.v2 had a limit of detection that was similar to an RT-qPCR cycle-threshold (Ct) value of 40.3 (Fig. S10D and S10E).
In blinded testing at an external laboratory at the University of Washington, we tested 202 SARS-CoV-2-positive and 200 SARS-CoV-2-negative nasopharyngeal swab samples obtained from patients. These samples were prepared by adding 50 μl of swab specimens obtained from patients with Covid-19 to a clean swab, in accordance with the recommendation of the Food and Drug Administration for simulating whole swabs for regulatory applications (see the Methods section in the Supplementary Appendix). This testing showed that STOPCovid.v2 had a sensitivity of 93.1% and a specificity of 98.5% (Figure 1B and 1C, Fig. S11A, and Table 1). STOPCovid.v2 false negative samples had RT-qPCR Ct values greater than 37. Positive samples were detected in 15 to 45 minutes. Finally, we used fresh, dry, anterior nasal swabs (collected according to the recommendations of the CDC) to validate STOPCovid.v2, and we correctly identified 5 positive samples (Ct values, 19 to 36) and 10 negative samples (Fig. S11B through S11E). A detailed protocol for STOPCovid.v2 is provided in the Supplementary Appendix. The simplified format of STOPCovid.V2 is suited for use in low-complexity clinical laboratories.
編集者へ:CRISPR(clustered regular interspaced short palindromic repeat)をベースとした診断検査は、ウイルスや細菌の病原体を検出するための新しいプラットフォームを提供する。SHERLOCK(specific high-sensitivity enzymatic reporter unlocking)のような方法は、通常、2段階のプロセス(標的増幅とCRISPR媒介核酸検出)を使用しており、SARS-CoV-2の検出に使用されてきた。しかしこれらのアプローチは、RNA抽出ステップと、複数の液体処理ステップを含むため、サンプルの二次汚染のリスクを高め、ポイントオブケア検査で使用されるものよりも複雑である。
ここでは、SARS-CoV-2の検出のための簡単な検査について述べる。この試験の感度は、逆転写-定量的ポリメラーゼ鎖反応(RT-qPCR)アッセイと同様である。STOP (SHERLOCK testing in one pot) は、簡略化されたウイルス RNA の抽出と、等温増幅と CRISPR による検出を組み合わせた合理化されたアッセイである。この試験は、1つの温度で1時間以内に、最小限の装置で行うことができる。
等温増幅とCRISPR媒介検出を統合するためには、両方のステップに対応できる共通の反応バッファーを開発する必要があった。ウイルスRNAを増幅するために、我々は逆転写とそれに続くループ媒介等温増幅(LAMP)を選択した。というのも、LAMP試薬は広く利用可能であり、Cas酵素に適合する一定のバッファーを使用しているからだ。LAMPは55〜70℃で動作し、Alicyclobacillus acidiphilus (AapCas12b)由来のCas12bのような耐熱性Cas酵素を必要とする。我々は、複数のLAMPプライマーセットおよびAapCas12bガイドRNA(ガイドRNAは、AapCas12bが標的DNAを認識し、切断するのを助ける)を系統的に評価し、ワンポット反応混合物中で、SARS-CoV-2ヌクレオカプシドタンパク質をコードする遺伝子Nを標的とするための最適な組み合わせを同定した(NEJM.orgで本文全文とともに入手可能な補足付録の図S1〜S3を参照のこと)。このアッセイをSTOPCovid, version 1 (STOPCovid.v1)と呼んだ。予想通り、LAMP単独では非特異的なシグナルを生成することができるのに対し、STOPCovid.v1検出は、標的が存在する場合にのみシグナルを生成した(図S3E)。STOPCovid.v1は、ラテラルフローおよび蛍光リードアウトと互換性があり、蛍光リードアウトを使用して内部コントロールを検出することができる(図S4〜S6)。
RNA抽出を簡略化し、感度を高めるために、磁気ビーズ精製法を適応させた(図S9)。磁気ビーズは、鼻咽頭全体または前鼻腔スワブからのSARS-CoV-2 RNAゲノムを1つのSTOPCovid反応混合物に濃縮した。溶解と磁気ビーズ結合のステップを組み合わせ、エタノール洗浄と溶出のステップを排除することで、サンプル抽出の時間を15分に短縮し、作業時間を最小限にして検査を合理化した。この合理化された試験を、STOPCovid, version 2 (STOPCovid.v2)(図1A)と呼ぶ。
我々は、STOPCovid.v2を、疾病管理予防センター(CDC)の標準的な2段階試験(すなわち、RNA抽出後にRT-qPCRを行う)と比較した(図S10C)。STOPCovid.v2の磁気ビーズによる基質濃度により、スワブサンプル全体からウイルスRNAを検出することができ、CDC試験の600倍のインプット(ウイルスRNA量)を得ることができた。その結果、STOPCovid.v2は、CDCの RT-qPCRテストで検出された30分の1のウイルス量を確実に検出した(サンプルあたり100コピー、または1ミリリットルあたり33コピーと比較して1ミリリットルあたり1000コピー)。鼻咽頭スワブサンプルからの2つの独立した希釈系列を分析したところ、STOPCovid.v2の検出限界は、RT-qPCRのサイクル閾値(Ct)値40.3に近いことが明らかになった(図S10DおよびS10E)。
ワシントン大学の外部研究室での盲検試験において、患者から得られた202の-CoV-2陽性検体および200のSARS-CoV-2陰性検体の鼻咽頭スワブサンプルを試験した。これらのサンプルは、規制申請のための全スワブをシミュレートするための食品医薬品局の勧告に従って、Covid-19を有する患者から得たスワブ検体50μlを清浄なスワブに添加することによって調製した(補足付録の「方法」の項を参照)。この試験により、STOPCovid.v2は93.1%の感度および98.5%の特異度を有することが示された(図1Bおよび1C、図S11A、および表1)。STOPCovid.v2偽陰性サンプルは、37より大きいRT-qPCR Ct値を有していた。陽性サンプルは、15〜45分で検出された。最後に、STOPCovid.v2を検証するために、新鮮で乾燥した前鼻腔スワブ(CDCの推奨に従って採取した)を使用し、5つの陽性サンプル(Ct値、19〜36)および10の陰性サンプルを正しく同定した(図S11B〜S11E)。STOPCovid.v2の詳細なプロトコルは付録に記載されている。STOPCovid.v2の簡易フォーマットは、複雑性の低い臨床検査室での使用に適している。
Figure2. STOPCovid, Version 2 (STOPCovid.v2) Test and Performance Evaluation.